Parmi les outils du génie génétique, on trouve des vecteurs (plasmides, phages, virus) et des enzymes.
I Les enzymes.
A Les exonucléases.
On trouve trois différents types d’exonucléase :- type I : elles coupent de 3’ –> 5’
- type II : elles coupent de 5’ –> 3’
- type III : elles coupent de 3’ –> 5’ et de 5’ –> 3’.
B Les endonucléases.
1 Principe de fonctionnement.
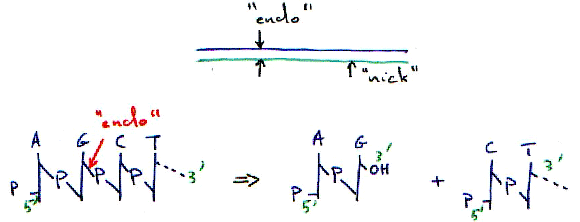
2 Les enzymes de restriction.
Une enzyme qui coupe à un endroit précis (toujours le même), sur un site de restriction est une enzyme spécifique : c’est une enzyme de restriction (ER). On trouve entre 300 et 350 ER.Les sites spécifiques sont en général constitués de 4pb (pour 200 ER), ou de 5pb (4 ou 5 ER) ou de 6pb (une trentaine d’ER) ou bien, de 7 à 12pb (quelques unes). Les enzymes de restriction utilisées sont celles qui reconnaissent un site (séquence) et coupe sur ce même site.
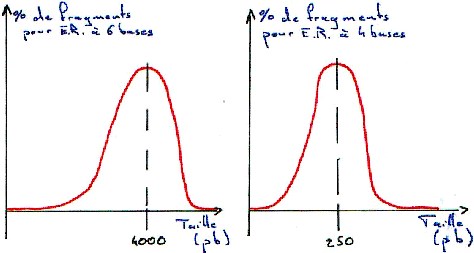
3 Digestion de l’ADN.
Si la digestion est complète, on obtiendra les résultant présentés dans les courbes suivantes. Dans ce cas, on se trouve en excès d’enzymes.Si la digestion n’est que partielle (ou ménagée), les fragments seront plus longs.
C La RNA polymérase II.
L’ARN pré-messager va donner un ARNm dans le cytosol. La famille des ARNu présente des cas petits, impliqués dans le processus de maturation des messagers.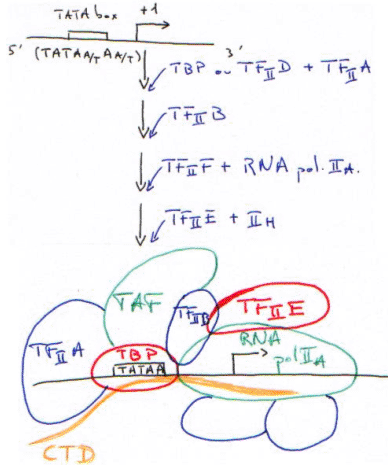
CTD : domaine carboxy-terminal de la grande sous-unité (220-240Da) de la RNA polymérase II.
Le TBP reconnaît la TATA-box puis recrute tous les autres facteurs.
La RNA polymérase II (14 sous-unités) interagit avec la TBP par l’intermédiaire du CTD.
Le complexe d’initiation est maintenant fermé.
Le CTD est représenté par 52 fois un heptapeptide (Y S P S T P S : tyrosine, sérine, proline, sérine, thréonine, proline, sérine). C’est un ensemble d’acides aminés phosphorylables par les enzymes sérine-thréonine-kinase et thyrosine-kinase.
Le CTD va se faire phosphoryler par un complexe enzymatique contenu dans TFIIA.
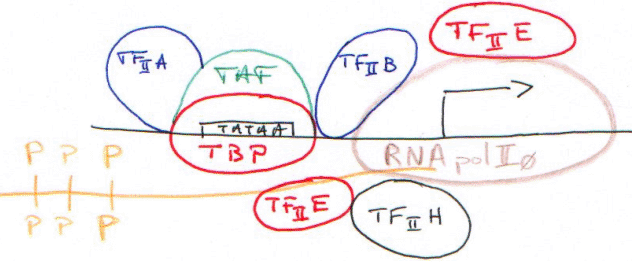
Quand le domaine carboxy-terminal est phosphorylé, on parle de RNApol II 0 : le CTD phosphorylé n’interagit plus avec la TBP.
TFIIH : ce facteur contient l’élément d’une maladie génétique : xéroderma pigmentosum, qui donne des difficultés à réparer les lésions sur l’ADN.
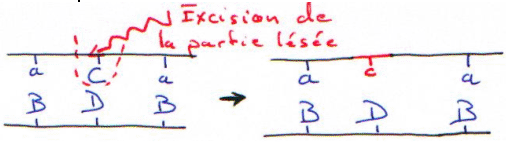
Dans cette maladie, l’activité de réparation est absente (maladie monogénique).
Ce facteur est composé de 9 sous-unités : c’est un facteur de transcription qui se comporte comme un facteur de réparation.
La kinase du CTD :
In vivo, en réalité, c’est un immense complexe préformé qui reconnaît le promoteur par l’intermédiaire du TBP qui reconnaît la TATA-box. Ce complexe est appelé une « holoenzyme ».
A ce complexe sont aussi associé les facteurs de maturation de l’ARNm. Ces facteurs sont reconnus par des TAF.
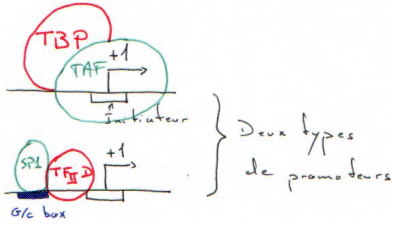
Les gènes possédant une G/C box sont appelés « gènes de ménage » ou « house keeping genes ».
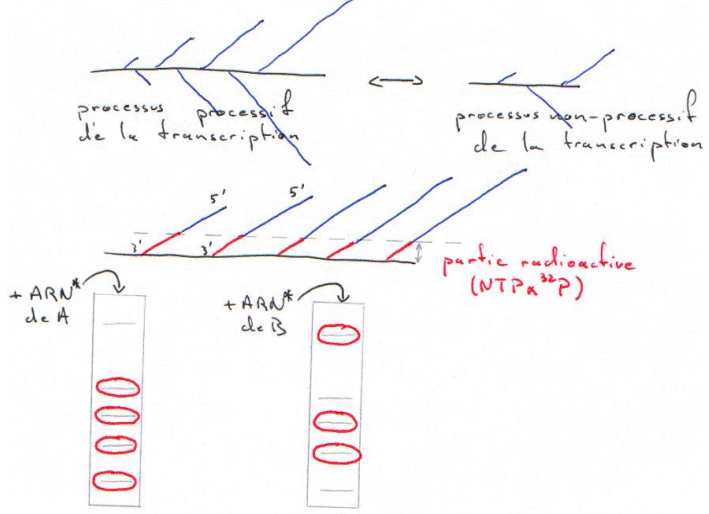
Exemple ; la technique de Run-On.
Cette technique repose sur le passage d’un processus non-processif de la transcription à un processus processif de la transcription. Lorsque l’on isole un noyau pour un gène donné, on fige son état transcriptionnel. In vivo, on lui fait reprendre la transcription (avec des NTP marqués).
On ne ré-initie pas la transcription de l’ARN, on ne fait qu’allonger des chaînes pré-initiées.
D La RNA polymérase III.
Dans une cellule, cette polymérase » transcrit des petits ARN : tRNA, ARN 5s et quelques uRNA.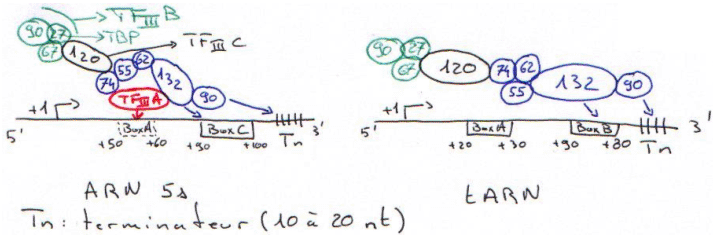
Dans le cas de l’ARN 5s, TFIIIA reconnaît une boite semblable à « boxA ».
TFIIIC recrute TFIIIB puis TBP. Ce dernier n’agit pas avec TCD.
Les RNA polymérases vont se servir d’une matrice ADN et utilisent 4 NTP (nucléotide tri-phosphate). Elles ont besoin d’un site de reconnaissance.
E Les DNAses ou RNAses : les nucléases.
1 Bal 31.
Cette enzyme attaque des deux côtés des brins Elle possède les deux activités : 5’3’nucléase et 3’5’nucléase.2 Nucléase S1.
Cette enzyme va dégrader l’ADN simple brin (et l’ARN).3 Nucléase micrococcus.
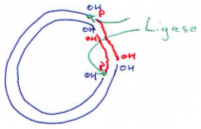
Cette enzyme digère aussi bien l’ADN que l’ARN.F La DNA ligase.
Cette enzyme est utilisée pour placer un insert dans un plasmide.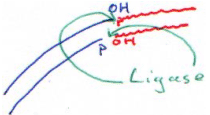
En général, on essaie de faire des coupures à extrémités cohésives. En fait, si l’on réalise une coupure qui donne des extrémités franches, la ligase doit rencontrer à la fois le plasmide et l’insert : il y a peu de probabilité de réussite.
- Se placer en excès de plasmides (100 fois plus).
- Eliminer la possibilité de re-fermeture du plasmide : on applique donc un traitement à la phosphatase.
- La fermeture du plasmide sur lui-même est impossible mais la ligation est toujours réalisable.
G La reverse transcriptase.
Dans un cas normal, on passe de l’ADN à l’ARN puis aux protéines. Ici, la matrice est l’ARN. La reverse transcriptase va incorporer des dNTP.Deux activités sont associées à cette enzyme :
- RNAse H : coupe l’hybride ADN/ARN.
- Terminal transférase : ajout de 3 à 5 Cytosine en 3’ si l’ARN en 5’ a subit le capping.
II Les problèmes de marquage de l’ADN.
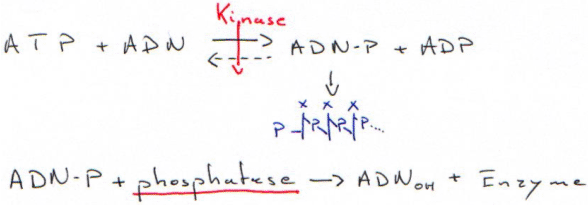
On peut marquer l’ADN avec une sonde radioactive (32P) ou avec une sonde fluorescente. Ce marquage est effectué soit aux extrémités de la molécule ou bien sur la totalité de celle-ci.On va utiliser des nucléotides au α32P pour marquer sur tout le long de la molécule ou bien des nucléotides au γ32P pour marquer uniquement les extrémités. Le marquage fluorescent se fait avec des bases fluorescentes.
1 Marquage terminal : le tailing.

2 Marquage terminal : nucléotide kinase et protéine kinase.
III Le séquençage et la PCR.
Il existe deux méthodes de séquençage : la méthode de Maxam et Gilbert et la méthode de Sanger.A La méthode de Maxam et Gilbert.
On prépare un tube par type de coupure (une après G, après A, après T, après C). En fonction des concentration d’enzyme et d’ADN, on obtient (statistiquement) une coupure par molécule avec tous les cas différents présents.B La méthode de Sanger.
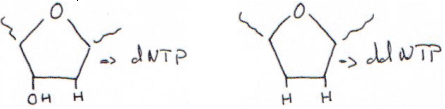
Dans ce cas, on va réaliser la synthèse d’ADN avec des arrêts de synthèse, en utilisant des didésoxynucléotides.L’amorce pour démarrer la synthèse est connue (et vient du commerce). On réalise ensuite la synthèse avec un ddNTP présent.
C La PCR.
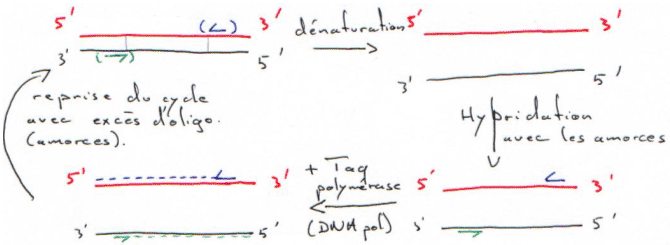
IV Construction d’une banque de cDNA.
En biologie moléculaire, les deux grandes problématiques sont : la structure des gènes et génomes, et, comment sont exprimés les différents gènes dans les divers types cellulaires.On va dégrader l’ADN par des ER afin d’obtenir des fragments homogènes en taille. On fait entrer ces fragments dans vecteurs, puis, dans des bactéries. On a alors une banque génomique : grand nombre de bactéries ayant intégré un fragment d’ADN (à cloner).
A Les vecteurs.
On a le choix entre différents vecteurs : phages et plasmides. En général, on va utiliser un phage spécial, le phage ? (50 000pb).1 Le phage λ.
Le « pili » de la queue du phage reconnaît la bactérie grâce aux récepteurs membranaires et va permettre l’injection de l’ADN dans cette bactérie.Cet ADN pourra servir dans deux cycles :
- Cycle lysogénique : l’ADN phagique s’insère dans le génome bactérien.
- Cycle lytique : il y a production massive de phages jusqu’à la lyse des bactéries.
Sur le phage λ, la partie permettant la lysogénie fait 15kb. Ce phage est composé d’un bras gauche de 10kb, du droit de 20kb et de la partie centrale « inutile » de 20kb.
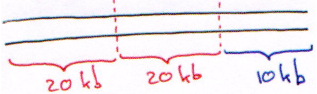
En réalité, on prend 2 ER et avec chacune d’elle, on effectue les digestions à 1/100ème. Dans ce cas, on aura une distribution homogène.
2 Les plasmides.
Les plasmides ont « Ori » et sont capables de se maintenir dans les bactéries. Leur taille est variable (de 4000pb à 10.000pb).En 1972, on a vu que les bactéries devaient être compétentes pour réaliser la « transformation bactérienne » : entrée d’ADN extérieur. Les facteurs de compétence sont le phosphate de calcium et la température.
Les plasmides ne seront pas utilisés pour les génomes de grande taille : le rendement de transformation diminue avec la taille (on a le maximum avec 10.000pb).
On va réaliser une combinaison entre le phage λ et un plasmide.
3 Les cosmides.
Les cosmides sont des vecteurs hybrides portant à la fois :- des séquences d’origine phagique permettant leur encapsidation in vitro (séquence cos).
- des séquences d’origine plasmidique : gène de résistance à un antibiotique et séquence permettant au plasmide de se répliquer dans une bactérie comme un simple s).
- des séquences d’origine plasmidique : gène de résistance à un antibiotique et séquence permettant au plasmide de se répliquer dans une bactérie comme un simple plasmide (séquence Ori).
Une fois que l’on a placé l’insert (fragment de 45kb), on réalise l’encapsidation et la mise en place de la queue.
Pour avoir un banque complète de cDNA , on prend 10 fois plus de bactéries que de nécessaire.
4 Les YAC (yeast artificial chromosome) ou chromosome de levure.
Ces vecteurs de clonages peuvent se présenter sous deux formes : une forme circulaire, permettant sa manipulation chez E. Coli (et en particulier l’insertion d’ADN exogène et son amplification –> ils possèdent une séquence ori et un gène de résistance à un antibiotique) et une forme linéaire, obtenue après coupure par BamHI et EcoRI et qui permet d’obtenir les deux “bras” du YAC. Ces deux bras contiennent les télomères (TEL), une origine de réplication levure (ARS) un centromère (CEN) ainsi que des gènes (URA et TRP) utilisés pour la sélection des clones. La ligation de ces bras à un très grand fragment d’ADN humain (100 à 1000 kb) extrait de cellules en culture le transforme en pseudo chromosome de levure capable après introduction dans ce microorganisme de se répliquer (grâce à ARS), de stabiliser ses extrémités (grâce aux séquences TEL) et de se répartir entre les cellules filles lors de la division cellulaire (grâce à la séquence CEN).Toutefois, ces YAC ont une affreuse tendance à se recombiner avec les chromosomes de la levure. En général, on ajoute entre 2.10^5 et 2.10^6pb.
B Banque de cDNA.
Dans un individu, on isole un type cellulaire et l’on focalise dessus. Pour l’homme, on a entre 20 et 30 cDNA différents.A partir d’ARNm polyA, on passe au cDNA, que l’on insère ensuite dans un vecteur lequel est transféré dans des bactéries.
1 La reverse transcriptase.
On va hybrider des oligodT (25) sur la queue polyA, c’est ensuite que l’on ajoute la reverse transcriptase et les dNTP. Une fois le travail de la reverse transcriptase achevé, on élimine l’ARN avec la nucléase S1.On transcrit enfin le brin d’ADN que l’on obtient, mais l’on n’a pas de cDNA pleine taille.
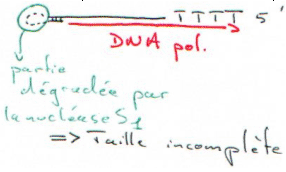
- RNAse H : elle crée des « nick » dans l’ARN mais avec l’extrémité 3’ portant un OH libre. L’hybride (ARN/ADN) obtenu aura donc un 3’ OH libre. Dans ce cas, on perd aussi un peu d’information en 5’. Le point fort est que l’on contrôle bien la réaction.
- Terminal transférase : On rajoute sur l’hybride un oligonucléotide avec trois G et 20 nucléotides. LA RNAse H va dégrader l’ARN mais pas l’amorce (l’oligo), ce qui permet de faire des cDNA pleine taille.

2 La mise en vecteur.
Les gènes donnent des messagers d’environ 3000nt (de 500 à 10^5nt). Dans ce cas, la taille est correcte pour une insertion dans un plasmide. On trouve deux types de plasmides :- Les « tout-bête » : ils possèdent une « Ori » et un MCS (pour les ER).
- Les plasmides « vecteurs d’expression » : ils ont une « ‘Ori », un MCS et un promoteur. Ils permettent la transcription des inserts, passage en mRNA et puis en protéine mais cette dernière étape est beaucoup plus dure. Pour qu’elle soit possible, il faut que le ribosome trouve un AUG et une séquence consensus (shine-dalgarno) en amont. On doit donc placer sur l’insert une séquence consensus et un AUG.
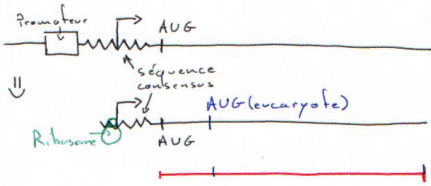
Pour le criblage d’expression, il vaut mieux utiliser des « tailles non entières ».
Dans les fibroblastes, on a environ 30.000 ARNm. On devrait prendre 30.000 bactéries mais on en prend plus car on n’a pas forcément une égalité de répartition des différents ARNm.
On va prendre 100 fois plus de bactéries, sinon, on peut faire des banques normalisées (avec la même quantité de chaque messager).
C Le criblage.
C’est dans l’étape de criblage où l’on fait du clonage.On étale la banque pour que chaque bactérie soit individualisée : elles donneront des cellules filles identiques (= clonage).
Pour repérer ce qui nous intéresse, il nous faut une sonde nucléotidique et une sonde Anticorps. On pourra identifier la protéine intéressante par spectrométrie de masse (de Malditoff ou de Cuthoff).
Pour la sonde anticorps, on fait faire à un animal, des anticorps contre une protéine (plus l’animal est gros, plus on récupère d’anticorps). Dans 50% des cas, on a des sondes hétérologues (sondes nucléotidiques mais trouvées dans une autre espèce).
La boite contenant les bactéries est répliquée sur un filtre de nitrate de cellulose. On lyse les bactéries répliquées et les macromolécules vont s’hybrider sur les filtres. On ajoute la sonde et on reconnaît (ou non) la présence de cDNA (s’il est présent). On récupère alors, sur la boite, la bactérie qui nous intéresse.
Avec un anticorps, on fait aussi une réplique sur nitrocellulose. La première réaction se fait avec des anticorps de lapin contre les protéines humaines (cDNA humaine). La seconde réaction consiste à rajouter un anticorps anti-lapin fluorescent contre l’anticorps de lapin.
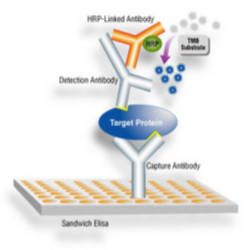
C’est une réaction élisa sandwich.
